ChIP-seq service
Die Chromatin-Immunpräzipitation (ChIP), eine klassische experimentelle Methode zur Untersuchung der Interaktion zwischen Protein und DNA, ist in verwandten Bereichen wie der Histonmodifikation und der durch spezifische Transkriptionsfaktoren regulierten Genexpression weit verbreitet. Mit der Entwicklung und Reife der NGS-Technologie ermöglicht die ChIP-Sequenzierung (ChIP-seq), eine Integration von Chromatin-Immunpräzipitationsexperimenten mit Hochdurchsatz-Sequenzierung, ein effizientes und genaues Screening von Proteinbindungsstellen im gesamten Genom. Darüber hinaus können DNA-Bindungsstellen, die mit spezialisierten Proteinen assoziiert sind, in verschiedenen Zelltypen und Geweben verglichen werden, da lebende Zellen aus Protein-DNA-Komplexen bestehen.
Einführung in die ChIP-Sequenzierung
ChIP-seq klingt wie RNA-seq geheimnisvoll und kompliziert, ist es aber nicht. Hier finden Sie eine sanfte Einführung in das Thema, die die Grundlagen des Experiments, die Verarbeitung der Daten und die Möglichkeiten, die Sie damit haben, erläutert.
Probenleitfaden
| Service | Probenart | Mindestmenge | Mindestkonzentration | Qualität |
|---|---|---|---|---|
| ChIP-seq | DNA | 10ng | 1 ng/µL | Fragmentiert auf 100-300bp |
*Die Messung der Konzentration mit Qubit wird dringend empfohlen.
*Bitte stellen Sie die Bioanalyzer-Ergebnisse für Ihre Probe zur Verfügung, falls diese vorhanden sind.
*Bitte liefern Sie die ChIP-DNA in nukleasefreiem Wasser. Wenn beim Betrieb des DNA-Chips in unserem Bioanalyzer 2100 RNase festgestellt wird, werden die Kosten für die Reinigung der durch RNase verursachten Kontamination in Rechnung gestellt.
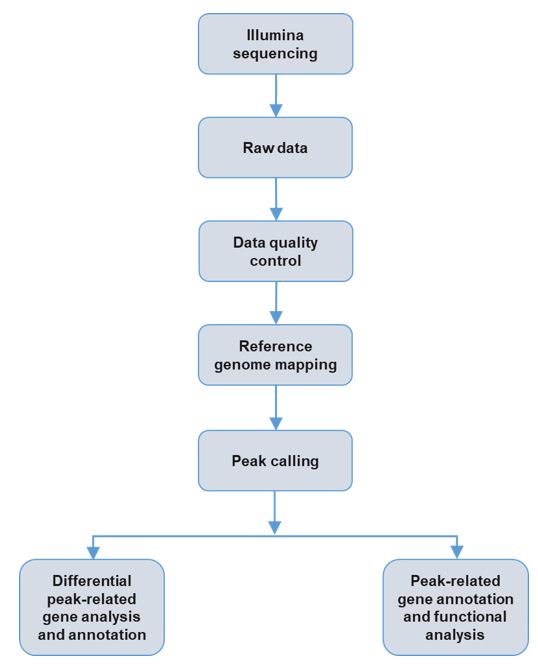
Service-Workflow
Analyse-Pipeline (allgemeiner Zweck)