Katsaus putkistoon
Bulk RNA-seq -putkisto kehitettiin osana ENCODE Uniform Processing Pipelines -sarjaa. Täydellinen putkikoodi on vapaasti saatavilla Githubissa, ja sitä voi käyttää DNAnexuksessa (linkki edellyttää tilin luomista) sen nykyisellä hinnoittelulla.
EnCODE Bulk RNA-seq -putkistoa voidaan käyttää sekä monistetuille että monistamattomille, parittaisille tai yksipäisille sekä säikeispesifisille tai ei-säikeispesifisille RNA-seq-kirjastoille. Kirjastot on tuotettava mRNA:sta (poly(A)+, rRNA:sta köyhdytetystä kokonais-RNA:sta tai poly(A)-populaatioista, joiden koko on valittu siten, että ne ovat pidempiä kuin noin 200 bp. Tulevaisuudessa tätä putkea voidaan käyttää myös PAS-seq- ja Bru-seq-datan käsittelyyn.
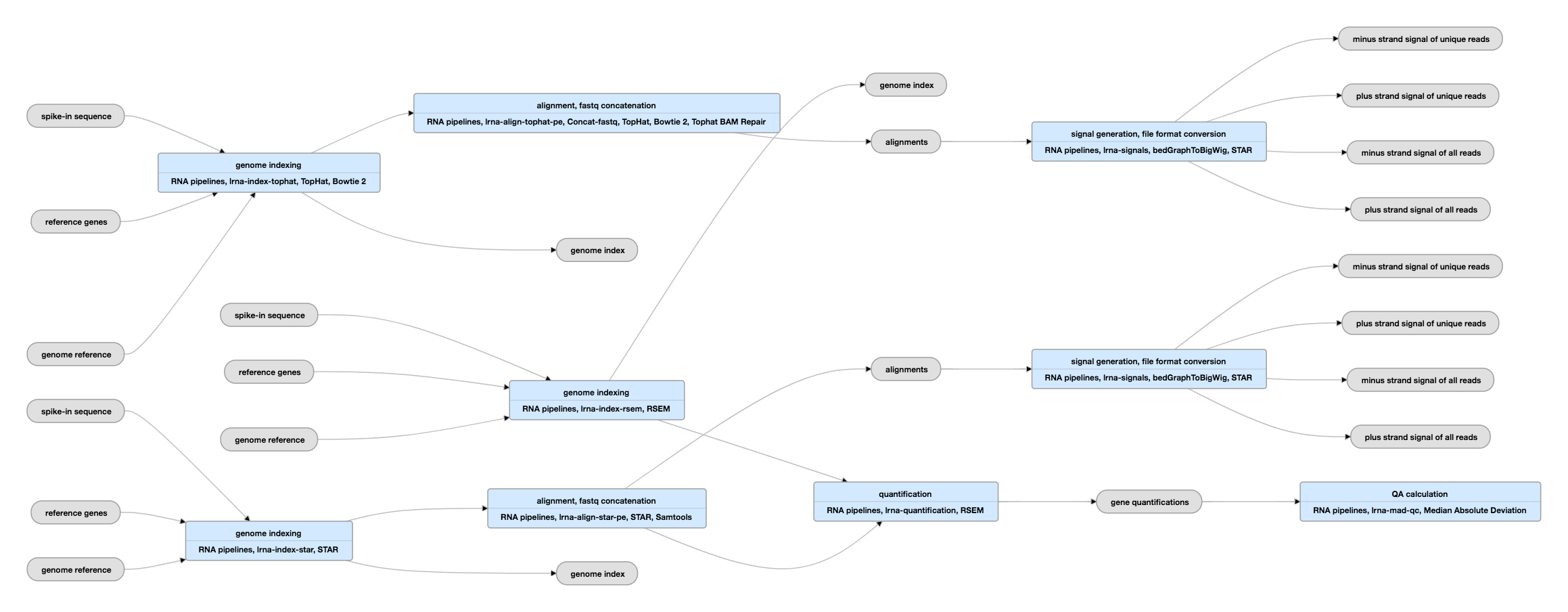
Putkilinjan kaavio pareittain erotetuille datoille
Katsele tämän putkilinjan tämänhetkistä instanssia pareittain erotetuille datoille
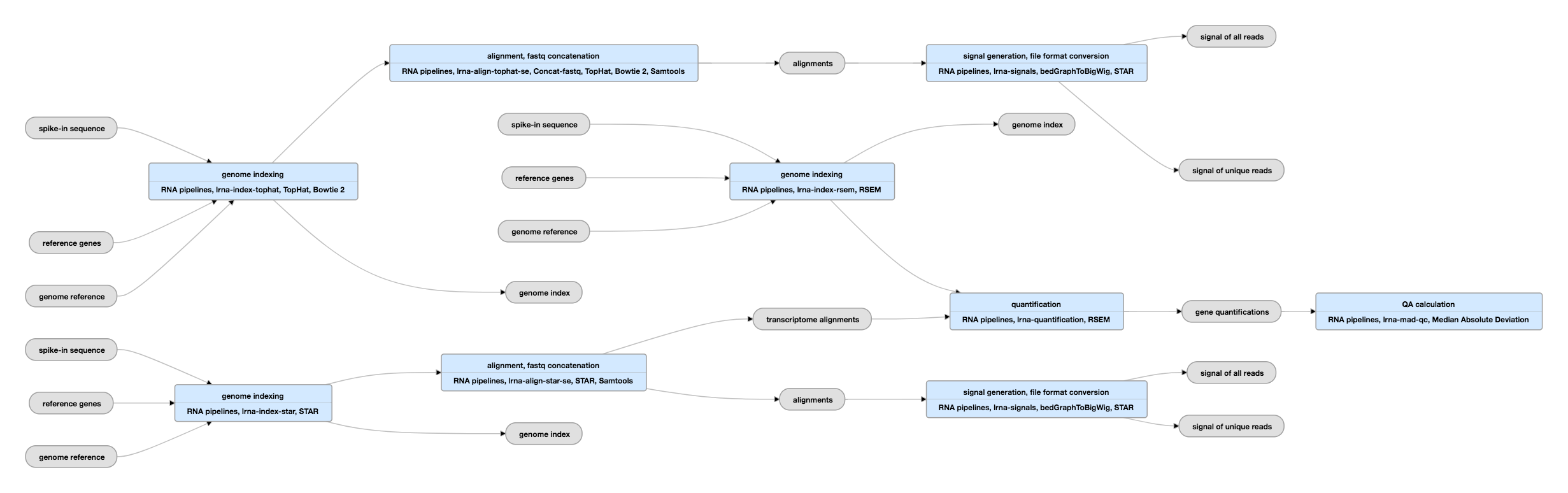
Putkilinjan kaavio yksittäispäätteisille datoille
Katsele tämän putkilinjan tämänhetkisiä instansseja yksittäispäätteisiä datoja varten
 >>
>>
Syötteet:
| Tiedostomuoto |
Tiedoston sisältämät tiedot |
tiedosto. description |
Notes |
| fastq |
reads |
G-zipped bulk RNA-seq reads | Lukujen on täytettävä Uniform Processing Pipeline Restrictions -kohdassa esitetyt kriteerit. |
| tar | genomi-indeksi | Generated by STAR or TopHat | Katsokaa ”Outputs”-taulukon alapuolella olevasta kohdasta ”Regarding alignment and quantification” (Kohdistus ja kvantitatiivisuus) lisätietoa kohdistusohjelmista (alignerit) ja niiden indekseistä. |
| fasta | spike-in-sekvenssi | ERCC Spike-ins (External RNA Control Consortium) | Spike-ins ovat käytännössä RNA-seq-kokeen kontrolleja. |
Tulokset:
| Tiedoston muoto |
Tiedoston sisältämät tiedot |
Tiedoston kuvaus |
Notes |
| bam | alignmentit | Tuotetaan kartoittamalla lukemat genomiin. | Katsokaa kohdasta ”Kohdistamisen ja kvantifioinnin osalta” ”Tuotokset”-taulukon alapuolella lisätietoja kohdistimista ja niiden indekseistä. |
| bam | transkriptomikohdistukset | Tuotetaan kartoittamalla lukuja transkriptomiin. | |
| bigWig | signal | Normalisoitu RNA-seq-signaali | Signaalit tuotetaan yksikäsitteisille lukusuureille ja yksikäsitteisille+multimapping-lukusuureille sekä plus- että miinussäikeissä. Ei-juosteiselle aineistolle signaalit luodaan uniikeille lukemille ja uniikeille+multimapping-lukemille ottamatta huomioon juosteen identiteettiä. |
| tsv | geenien kvantifioinnit | Sisältää spike-ins-kvantifioinnit |
Tiedostomuodon spesifikaatiot ovat seuraavat:
|
| tsv | transkriptien kvantifioinnit | Sisältää spike-insin kvantifioinnit | Katsokaa transkriptien kvantifiointeja koskeva varoitus alla olevassa kappaleessa otsikolla ”Kohdistamisen ja kvantifioinnin osalta”. |
| Putkisto tuottaa myös laatumittareita, kuten Spearmanin korrelaatio ja lukusyvyys. | |||
Kohdistamisesta ja kvantifioinnista:
Lukulukujen kartoitus tehdään STAR-ohjelmalla (joissakin tapauksissa käytetään sekä STAR- että TopHat-alignaattoreita erillisten bam-tiedostojen tuottamiseksi) ja geenien ja transkriptien kvantifiointi tehdään RSEM-ohjelmalla. Vaikka eri RNA-seq-putkistojen tuottamat kartoitukset ja geenien kvantifioinnit ovat yleisesti ottaen yhteneväisiä, yksittäisten transkriptien isoformien kvantifioinnit ovat paljon monimutkaisempia, ja ne voivat vaihdella huomattavasti käytetystä prosessointiputkistosta riippuen, eikä niiden tarkkuutta tunneta. Siksi kohdistuksia ja geenien kvantifiointeja voidaan käyttää luottavaisin mielin, kun taas transkriptien kvantifiointeja on käytettävä varoen.
Genomiviitteet
Katsele tässä putkistossa käytettyjä genomiviitteitä ja kromosomikokoja
Nämä putkistot vaativat sekä kokoonpanotietoja kiinnostuksen kohteena olevasta lajista että geeniviitteen. Kukin pääohjelmista, TopHat, STAR ja RSEM, luo indeksin käytettäväksi seuraavissa vaiheissa. Lisätietoja RSEM:n käytöstä on saatavilla täältä.
Exogeeniset RNA-spike-in-kontrollit
Exogeeniset RNA-spike-in-kontrollit lisätään näytteisiin vakiomuotoisen perusviivan luomiseksi RNA-ekspression kvantifiointia varten (PMC3166838). ENCODE-konsortio standardoi käyttämään kaupallisesti saatavilla olevia Ambion Mix 1 -spike-in-kontrolleja laimennoksella, joka on ~2 % lopullisista kartoitetuista lukemista. Käytössä on kuitenkin sekoitus vanhempaa dataa ja tuotua dataa. Siksi tietyssä kirjastossa käytettyjen spike-inien seuraamiseksi kirjastoon on liitetty tietokokonaisuus. Kyseinen tietokokonaisuus sisältää spike-ins-sekvenssitiedoston fasta-muodossa ja tiedot konsentraatioista. Näiden spike-in-sekvenssien odotetaan löytyvän kartoitusvaiheessa (-vaiheissa) käytetystä genomi-indeksistä ja myöhemmin luodusta bam-tiedostosta. Sekvenssien kvantifioinnit löytyvät RSEM:n transkripti- ja geenikvantifiointitiedostoista.
Katsele spike-ins-tietoaineistoja
Katsele ERCC:n spike-ins-analyysin todistusta
KäyTTää ERCC:n dash boardia
Linkit ja julkaisut
Löydä tällä putkiputkiprosessilla tuotettuja tietoja:
Tutustu julkaisuihin (tekeillä)
Katsele julkaisuja (työn alla)
