- Séquençage du génome. Séquençage
- Caractérisation génétique
- Méthodes de séquençage du génome de la grippe
Séquençage du génome
Les virus de la grippe changent constamment, en fait, tous les virus de la grippe subissent des modifications génétiques au fil du temps (pour plus d’informations, voir Comment le virus de la grippe peut changer : « Drift » et « Shift »). Le génome d’un virus grippal est constitué de tous les gènes qui composent le virus. Les CDC effectuent une surveillance tout au long de l’année des virus grippaux en circulation afin de suivre les modifications du génome (ou de parties du génome) de ces virus. Ce travail est effectué dans le cadre de la surveillance de routine de la grippe aux États-Unis et dans le cadre du rôle du CDC en tant que centre collaborateur de l’Organisation mondiale de la santé (OMS) pour la référence et la recherche sur la grippe. Les informations que les CDC recueillent en étudiant les modifications génétiques (également appelées « substitutions », « variantes » ou « mutations ») des virus de la grippe jouent un rôle important en matière de santé publique en aidant à déterminer si les vaccins et les médicaments antiviraux seront efficaces contre les virus de la grippe actuellement en circulation, ainsi qu’en aidant à déterminer le potentiel des virus de la grippe chez les animaux à infecter les humains.
Le séquençage du génome révèle la séquence des nucléotides dans un gène, comme les lettres de l’alphabet dans les mots. Les nucléotides sont des molécules organiques qui constituent l’unité structurelle de base des acides nucléiques, tels que l’ARN ou l’ADN. Tous les virus de la grippe sont constitués d’ARN simple brin, par opposition à l’ADN double brin. Les gènes ARN des virus de la grippe sont constitués de chaînes de nucléotides liées entre elles et codées par les lettres A, C, G et U, qui signifient respectivement adénine, cytosine, guanine et uracile. La comparaison de la composition des nucléotides dans un gène de virus avec l’ordre des nucléotides dans un gène de virus différent peut révéler des variations entre les deux virus.
Les variations génétiques sont importantes car elles peuvent affecter la structure des protéines de surface d’un virus grippal. Les protéines sont constituées de séquences d’acides aminés.
La substitution d’un acide aminé par un autre peut affecter les propriétés d’un virus, comme le degré de transmission du virus entre les personnes, et la sensibilité du virus aux médicaments antiviraux ou aux vaccins actuels.


Le séquençage du génome révèle la séquence des nucléotides dans un gène, comme les lettres de l’alphabet dans les mots. Comparer la composition des nucléotides dans un gène de virus avec l’ordre des nucléotides dans un gène de virus différent peut révéler des variations entre les deux virus.
Les variations génétiques sont importantes car elles affectent la structure des protéines de surface d’un virus de la grippe. Les protéines sont constituées de séquences d’acides aminés.
La substitution d’un acide aminé par un autre peut affecter les propriétés d’un virus, comme le degré de transmission entre les personnes et la sensibilité du virus aux médicaments antiviraux ou aux vaccins actuels.
Les virus de la grippe A et B – les principaux virus de la grippe qui infectent les personnes – sont des virus à ARN qui possèdent huit segments de gènes. Ces gènes contiennent des « instructions » pour fabriquer de nouveaux virus, et ce sont ces instructions qu’un virus de la grippe utilise une fois qu’il a infecté une cellule humaine pour tromper la cellule et l’amener à produire d’autres virus de la grippe, propageant ainsi l’infection.
Les gènes de la grippe sont constitués d’une séquence de molécules appelées nucléotides qui se lient entre elles sous forme de chaîne. Les nucléotides sont désignés par les lettres A, C, G et U.
Le séquençage du génome est un processus qui détermine l’ordre, ou séquence, des nucléotides (c’est-à-dire A, C, G et U) dans chacun des gènes présents dans le génome du virus. Le séquençage du génome complet peut révéler la séquence d’environ 13 500 lettres de tous les gènes du génome du virus.
Chaque année, le CDC effectue le séquençage du génome complet d’environ 7 000 virus de la grippe à partir d’échantillons cliniques originaux recueillis dans le cadre de la surveillance virologique. Le génome d’un virus grippal A ou B contient huit segments de gènes qui codent (c’est-à-dire déterminent la structure et les caractéristiques) les 12 protéines du virus, y compris ses deux principales protéines de surface : l’hémagglutinine (HA) et la neuraminidase (NA). Les protéines de surface d’un virus de la grippe déterminent des propriétés importantes du virus, notamment la façon dont le virus réagit à certains médicaments antiviraux, la similarité génétique du virus avec les virus actuels du vaccin contre la grippe et le potentiel des virus de la grippe zoonotiques (d’origine animale) à infecter des hôtes humains.
Caractérisation génétique
Le CDC et d’autres laboratoires de santé publique dans le monde entier séquencent les gènes des virus de la grippe depuis les années 1980. Le CDC fournit des séquences génétiques à des bases de données publiques, telles que GenBankexternal icon et la Global Initiative on Sharing Avian Influenza Data (GISAID)external icon, à l’usage des chercheurs en santé publique. Les bibliothèques de séquences génétiques qui en résultent permettent au CDC et à d’autres laboratoires de comparer les gènes des virus de la grippe actuellement en circulation avec les gènes des anciens virus de la grippe et des virus utilisés dans les vaccins. Ce processus de comparaison des séquences génétiques est appelé caractérisation génétique. Le CDC utilise la caractérisation génétique pour les raisons suivantes :
- Déterminer à quel point les virus de la grippe sont génétiquement « apparentés » ou similaires les uns aux autres
- Suivre l’évolution des virus de la grippe
- Identifier les changements génétiques qui affectent les propriétés du virus. Par exemple, pour identifier les changements spécifiques qui sont associés aux virus de la grippe se propageant plus facilement, provoquant des maladies plus graves ou développant une résistance aux médicaments antiviraux
- Pour évaluer dans quelle mesure un vaccin contre la grippe pourrait protéger contre un virus de la grippe particulier en fonction de sa similarité génétique avec le virus
- Pour surveiller les changements génétiques des virus de la grippe circulant dans les populations animales qui pourraient leur permettre d’infecter les humains.
Les différences relatives entre un groupe de virus de la grippe sont montrées en les organisant dans un graphique appelé « arbre phylogénétique ». Les arbres phylogénétiques pour les virus de la grippe sont comme des arbres de famille (généalogie) pour les gens. Ces arbres montrent à quel point les virus individuels sont « apparentés » les uns aux autres. Les virus sont regroupés selon que les nucléotides de leurs gènes sont identiques ou non. Les arbres phylogénétiques des virus de la grippe montrent généralement dans quelle mesure les gènes de l’hémagglutinine (HA) ou de la neuraminidase (NA) des virus sont similaires les uns aux autres. Chaque séquence d’un virus grippal spécifique possède sa propre branche sur l’arbre. Le degré de différence génétique (nombre de différences nucléotidiques) entre les virus est représenté par la longueur des lignes horizontales (branches) de l’arbre phylogénétique. Plus les virus sont éloignés sur l’axe horizontal d’un arbre phylogénétique, plus les virus sont génétiquement différents les uns des autres.
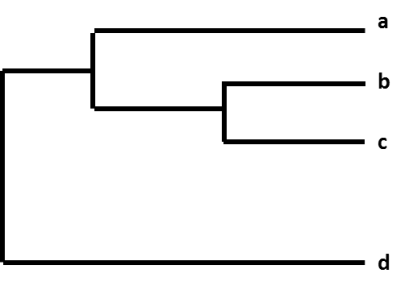
Figure. Un arbre phylogénétique.
Par exemple, après que le CDC ait séquencé un virus de la grippe A(H3N2) collecté dans le cadre de la surveillance, la séquence du virus est cataloguée avec d’autres séquences de virus qui ont un gène HA similaire (H3), et un gène NA similaire (N2). Dans le cadre de ce processus, les CDC comparent la nouvelle séquence virale avec les autres séquences virales et recherchent les différences entre elles. Le CDC utilise ensuite un arbre phylogénétique pour représenter visuellement à quel point les virus A(H3N2) sont génétiquement différents les uns des autres.
Le CDC effectue une caractérisation génétique des virus de la grippe tout au long de l’année. Ces données génétiques sont utilisées conjointement avec les données de caractérisation antigénique des virus pour aider à déterminer quels virus vaccinaux devraient être choisis pour les prochains vaccins contre la grippe de l’hémisphère nord ou de l’hémisphère sud. Dans les mois qui précèdent les réunions de consultation de l’OMS sur les vaccins, en février et en septembre, les CDC collectent des virus de la grippe par le biais de la surveillance et comparent les séquences des gènes HA et NA des virus vaccinaux actuels avec celles des virus de la grippe en circulation. C’est une façon d’évaluer le degré de parenté entre les virus de la grippe en circulation et les virus contre lesquels le vaccin contre la grippe saisonnière a été formulé. Au fur et à mesure que les virus sont collectés et caractérisés génétiquement, des différences peuvent être révélées.
Par exemple, parfois, au cours d’une saison, les virus circulants changent génétiquement, ce qui fait qu’ils deviennent différents du virus vaccinal correspondant. C’est une indication qu’un virus vaccinal différent peut devoir être sélectionné pour le vaccin de la saison grippale suivante, bien que d’autres facteurs, notamment les résultats de la caractérisation antigénique, influencent fortement les décisions relatives aux vaccins. Les protéines de surface HA et NA des virus de la grippe sont des antigènes, ce qui signifie qu’elles sont reconnues par le système immunitaire et capables de déclencher une réponse immunitaire, notamment la production d’anticorps qui peuvent bloquer l’infection. La caractérisation antigénique fait référence à l’analyse de la réaction d’un virus avec les anticorps pour aider à évaluer comment il se rapporte à un autre virus.
Méthodes de séquençage du génome de la grippe
Un échantillon de grippe contient de nombreuses particules de virus de la grippe qui ont été cultivées dans un tube à essai et qui présentent souvent de petites différences génétiques les unes par rapport aux autres dans l’ensemble de la population de virus frères et sœurs.
Traditionnellement, les scientifiques ont utilisé une technique de séquençage appelée « la réaction de Sanger » pour suivre l’évolution de la grippe dans le cadre de la surveillance virologique. Le séquençage de Sanger identifie la séquence génétique prédominante parmi les nombreux virus de la grippe présents dans un isolat. Cela signifie que les petites variations dans la population de virus présents dans un échantillon ne sont pas reflétées dans le résultat final. Les scientifiques utilisent souvent la méthode Sanger pour effectuer un séquençage partiel du génome des virus de la grippe, alors que les technologies plus récentes (voir paragraphe suivant) sont mieux adaptées au séquençage du génome entier.
Au cours des cinq dernières années, le CDC a utilisé des méthodologies de « séquençage de nouvelle génération (NGS) », qui ont considérablement augmenté la quantité d’informations et de détails que l’analyse de séquençage peut fournir. Le NGS utilise la détection moléculaire avancée (DMA) pour identifier les séquences génétiques de chaque virus dans un échantillon. Par conséquent, la NGS révèle les variations génétiques entre de nombreuses particules différentes du virus de la grippe dans un seul échantillon, et ces méthodes révèlent également la région codante entière des génomes. Ce niveau de détail peut directement profiter aux décisions de santé publique de manière importante, mais les données doivent être soigneusement interprétées par des experts hautement qualifiés dans le contexte des autres informations disponibles. Voir les projets AMD : Improving Influenza Vaccines pour plus d’informations sur la façon dont le NGS et l’AMD révolutionnent la cartographie du génome de la grippe au CDC.
