ChIP-seq service
Chromatine-immunoprecipitatie (ChIP), een klassieke experimentele methode voor het bestuderen van de interactie tussen eiwit en DNA, wordt veel gebruikt op verwante gebieden zoals histonmodificatie en genexpressie die door specifieke transcriptiefactoren wordt gereguleerd. Met de ontwikkeling en maturiteit van NGS technologie, maakt ChIP sequenering (ChIP-seq), een integratie van chromatine immunoprecipitatie experimenten met high-throughput sequenering, efficiënte en nauwkeurige screening van eiwit-bindende sites over het genoom mogelijk. Bovendien kunnen DNA-bindende sites geassocieerd met gespecialiseerde eiwitten worden vergeleken in verschillende celtypes en weefsels als gevolg van de levende cellen ontstaan in eiwit-DNA complexen.
Intro to ChIP Sequencing
ChIP-seq, net als RNA-seq, klinkt mysterieus en ingewikkeld, maar dat is het niet. Hier is een inleiding tot het onderwerp dat de basisprincipes van het experiment behandelt, hoe de gegevens worden verwerkt en wat je er allemaal mee kunt doen.
Sample richtlijn
| Service | Soort monster | Minimale hoeveelheid | Minimale concentratie | Kwaliteit |
|---|---|---|---|---|
| ChIP-seq | DNA | 10ng | 1 ng/µL | Gefragmenteerd tot 100-300bp |
*Het meten van de concentratie met Qubit wordt ten zeerste aanbevolen.
*Gelieve de resultaten van de Bioanalyzer voor uw monster mee te delen indien deze beschikbaar zijn.
*Gelieve het ChIP-afgeleide DNA in nucleasevrij water te leveren. Als bij het uitvoeren van de DNA-chip in onze Bioanalyzer 2100 RNase wordt aangetroffen, worden de kosten voor het opruimen van de door RNase veroorzaakte verontreiniging in rekening gebracht.
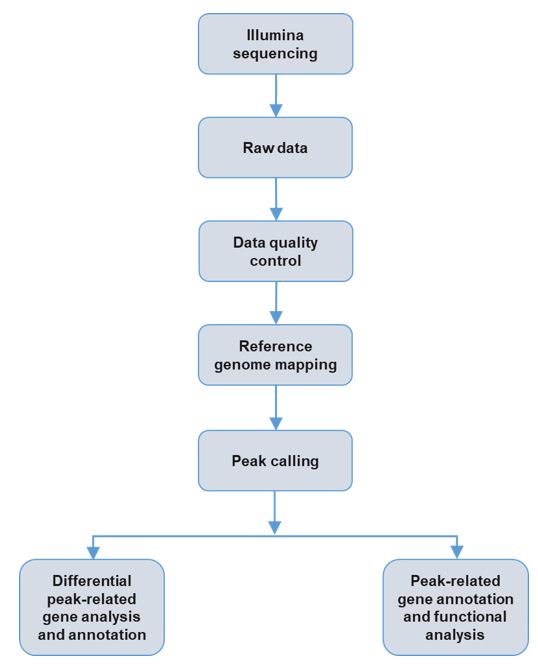
Service workflow
Analysepijplijn (algemeen gebruik)