- Secuenciación del genoma Secuenciación
- Caracterización genética
- Métodos de secuenciación del genoma de la gripe
Secuenciación del genoma
Los virus de la gripe cambian constantemente, de hecho, todos los virus de la gripe sufren cambios genéticos a lo largo del tiempo (para más información, consulte Cómo puede cambiar el virus de la gripe: «Drift» y «Shift»). El genoma de un virus de la gripe está formado por todos los genes que componen el virus. Los CDC realizan una vigilancia durante todo el año de los virus de la gripe en circulación para controlar los cambios en el genoma (o en partes del genoma) de estos virus. Este trabajo se realiza como parte de la vigilancia rutinaria de la influenza en los Estados Unidos y como parte del papel de los CDC como Centro Colaborador de la Organización Mundial de la Salud (OMS) para la Referencia e Investigación de la Influenza. La información que los CDC recopilan a partir del estudio de los cambios genéticos (también conocidos como «sustituciones», «variantes» o «mutaciones») en los virus de la influenza desempeña un importante papel en la salud pública, ya que ayuda a determinar si las vacunas y los medicamentos antivirales funcionarán contra los virus de la influenza que circulan actualmente, además de ayudar a determinar el potencial de los virus de la influenza en animales para infectar a los seres humanos.
La secuenciación del genoma revela la secuencia de los nucleótidos en un gen, como las letras del alfabeto en las palabras. Los nucleótidos son moléculas orgánicas que forman la unidad estructural de los ácidos nucleicos, como el ARN o el ADN. Todos los virus de la gripe están formados por ARN monocatenario, a diferencia del ADN bicatenario. Los genes de ARN de los virus de la gripe están formados por cadenas de nucleótidos unidos entre sí y codificados por las letras A, C, G y U, que significan adenina, citosina, guanina y uracilo, respectivamente. La comparación de la composición de los nucleótidos de un gen del virus con el orden de los nucleótidos de otro gen del virus puede revelar variaciones entre los dos virus.
Las variaciones genéticas son importantes porque pueden afectar a la estructura de las proteínas de superficie de un virus de la gripe. Las proteínas están formadas por secuencias de aminoácidos.
La sustitución de un aminoácido por otro puede afectar a las propiedades de un virus, como la capacidad de transmisión entre personas y la susceptibilidad del virus a los medicamentos antivirales o a las vacunas actuales.


La secuenciación del genoma revela la secuencia de los nucleótidos de un gen, como las letras del alfabeto en las palabras. La comparación de la composición de los nucleótidos de un gen del virus con el orden de los nucleótidos de otro gen del virus puede revelar variaciones entre los dos virus.
Las variaciones genéticas son importantes porque afectan a la estructura de las proteínas de superficie de un virus de la gripe. Las proteínas están formadas por secuencias de aminoácidos.
La sustitución de un aminoácido por otro puede afectar a las propiedades de un virus, como la capacidad de transmisión entre personas y la susceptibilidad del virus a los fármacos antivirales o a las vacunas actuales.
Los virus de la gripe A y B -los principales virus de la gripe que infectan a las personas- son virus de ARN que tienen ocho segmentos genéticos. Estos genes contienen «instrucciones» para fabricar nuevos virus, y son estas instrucciones las que un virus de la gripe utiliza una vez que infecta una célula humana para engañar a la célula para que produzca más virus de la gripe, propagando así la infección.
Los genes de la gripe están formados por una secuencia de moléculas llamadas nucleótidos que se unen en forma de cadena. Los nucleótidos se designan con las letras A, C, G y U.
La secuenciación del genoma es un proceso que determina el orden, o la secuencia, de los nucleótidos (es decir, A, C, G y U) en cada uno de los genes presentes en el genoma del virus. La secuenciación del genoma completo puede revelar la secuencia de aproximadamente 13.500 letras de todos los genes del genoma del virus.
Cada año los CDC realizan la secuenciación del genoma completo de unos 7.000 virus de la gripe a partir de muestras clínicas originales recogidas mediante la vigilancia virológica. El genoma de los virus de la gripe A o B contiene ocho segmentos de genes que codifican (es decir, determinan la estructura y las características) de las 12 proteínas del virus, incluidas sus dos principales proteínas de superficie: la hemaglutinina (HA) y la neuraminidasa (NA). Las proteínas de superficie de un virus de la gripe determinan propiedades importantes del virus, incluyendo la forma en que el virus responde a ciertos medicamentos antivirales, la similitud genética del virus con los actuales virus de la vacuna contra la gripe y el potencial de los virus de la gripe zoonótica (de origen animal) para infectar a los huéspedes humanos.
Caracterización genética
El CDC y otros laboratorios de salud pública de todo el mundo han estado secuenciando los genes de los virus de la gripe desde la década de 1980. Los CDC contribuyen con secuencias genéticas a bases de datos públicas, como GenBankexternal icon y la Global Initiative on Sharing Avian Influenza Data (GISAID)external icon, para su uso por parte de los investigadores de salud pública. Las bibliotecas de secuencias genéticas resultantes permiten a los CDC y a otros laboratorios comparar los genes de los virus de la gripe que circulan actualmente con los genes de los virus de la gripe más antiguos y con los virus utilizados en las vacunas. Este proceso de comparación de secuencias genéticas se denomina caracterización genética. Los CDC utilizan la caracterización genética por los siguientes motivos:
- Para determinar cuán estrechamente «relacionados» o similares son los virus de la gripe desde el punto de vista genético
- Para monitorear cómo evolucionan los virus de la gripe
- Para identificar los cambios genéticos que afectan las propiedades del virus. Por ejemplo, para identificar los cambios específicos que se asocian a los virus de la gripe que se propagan con mayor facilidad, que causan una enfermedad más grave o que desarrollan resistencia a los fármacos antivirales
- Para evaluar la eficacia de una vacuna contra la gripe que podría proteger contra un determinado virus de la gripe en función de su similitud genética con el virus
- Para vigilar los cambios genéticos en los virus de la gripe que circulan en las poblaciones animales que podrían permitirles infectar a los seres humanos.
Las diferencias relativas entre un grupo de virus de la gripe se muestran organizándolas en un gráfico llamado «árbol filogenético». Los árboles filogenéticos de los virus de la gripe son como los árboles genealógicos de las personas. Estos árboles muestran el grado de «parentesco» entre los distintos virus. Los virus se agrupan en función de si los nucleótidos de sus genes son idénticos o no. Los árboles filogenéticos de los virus de la gripe suelen mostrar el grado de similitud de los genes de la hemaglutinina (HA) o la neuraminidasa (NA) de los virus. Cada secuencia de un virus de la gripe específico tiene su propia rama en el árbol. El grado de diferencia genética (número de diferencias de nucleótidos) entre los virus está representado por la longitud de las líneas horizontales (ramas) en el árbol filogenético. Cuanto más separados estén los virus en el eje horizontal de un árbol filogenético, más diferentes genéticamente son los virus entre sí.
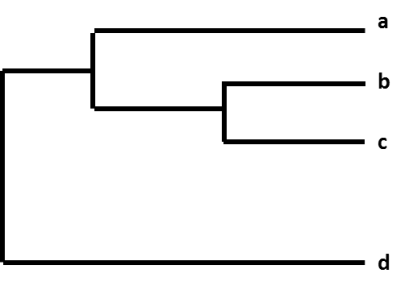
Figura. Un árbol filogenético.
Por ejemplo, después de que los CDC secuencien un virus de la gripe A(H3N2) recogido a través de la vigilancia, la secuencia del virus se cataloga con otras secuencias de virus que tienen un gen HA similar (H3), y un gen NA similar (N2). Como parte de este proceso, los CDC comparan la nueva secuencia del virus con las otras secuencias del virus y buscan diferencias entre ellas. A continuación, los CDC utilizan un árbol filogenético para representar visualmente las diferencias genéticas entre los virus A(H3N2).
Los CDC realizan la caracterización genética de los virus de la gripe durante todo el año. Estos datos genéticos se utilizan junto con los datos de caracterización antigénica de los virus para ayudar a determinar qué virus vacunales deben elegirse para las próximas vacunas contra la gripe del hemisferio norte o del hemisferio sur. En los meses previos a las reuniones de consulta sobre vacunas de la OMS, que se celebran en febrero y septiembre, los CDC recogen virus de la gripe a través de la vigilancia y comparan las secuencias genéticas de la HA y la NA de los actuales virus de la vacuna con las de los virus de la gripe en circulación. Esta es una forma de evaluar el grado de parentesco de los virus de la gripe en circulación con los virus contra los que se formuló la vacuna contra la gripe estacional. A medida que se recogen los virus y se caracterizan genéticamente, se pueden revelar las diferencias.
Por ejemplo, a veces en el transcurso de una temporada, los virus circulantes cambian genéticamente, lo que hace que se diferencien del correspondiente virus de la vacuna. Este es un indicio de que puede ser necesario seleccionar un virus vacunal diferente para la vacuna de la siguiente temporada de gripe, aunque otros factores, incluidos los resultados de la caracterización antigénica, influyen en gran medida en las decisiones sobre la vacuna. Las proteínas de superficie HA y NA de los virus de la gripe son antígenos, lo que significa que son reconocidos por el sistema inmunitario y son capaces de desencadenar una respuesta inmunitaria, incluida la producción de anticuerpos que pueden bloquear la infección. La caracterización antigénica se refiere al análisis de la reacción de un virus con los anticuerpos para ayudar a evaluar cómo se relaciona con otro virus.
Métodos de secuenciación del genoma de la gripe
Una muestra de gripe contiene muchas partículas de virus de la gripe que se cultivaron en un tubo de ensayo y que a menudo tienen pequeñas diferencias genéticas en comparación entre toda la población de virus hermanos.
Tradicionalmente, los científicos han utilizado una técnica de secuenciación llamada «reacción de Sanger» para controlar la evolución de la gripe como parte de la vigilancia virológica. La secuenciación de Sanger identifica la secuencia genética predominante entre los numerosos virus de la gripe que se encuentran en un aislado. Esto significa que las pequeñas variaciones en la población de virus presentes en una muestra no se reflejan en el resultado final. Los científicos suelen utilizar el método Sanger para llevar a cabo la secuenciación parcial del genoma de los virus de la gripe, mientras que las tecnologías más recientes (véase el siguiente párrafo) son más adecuadas para la secuenciación del genoma completo.
En los últimos cinco años, los CDC han utilizado metodologías de «secuenciación de próxima generación (NGS)», que han ampliado en gran medida la cantidad de información y los detalles que puede proporcionar el análisis de secuenciación. La NGS utiliza la detección molecular avanzada (AMD) para identificar las secuencias genéticas de cada virus en una muestra. Por lo tanto, la NGS revela las variaciones genéticas entre muchas partículas diferentes del virus de la gripe en una sola muestra, y estos métodos también revelan toda la región de codificación de los genomas. Este nivel de detalle puede beneficiar directamente la toma de decisiones en materia de salud pública de manera importante, pero los datos deben ser interpretados cuidadosamente por expertos altamente capacitados en el contexto de otra información disponible. Véase Proyectos AMD: Improving Influenza Vaccines para obtener más información sobre cómo la NGS y la AMD están revolucionando el mapeo del genoma de la gripe en los CDC.
