Un pulmón humano enfermo, fijado en el conservante formol durante más de 100 años, ayudó a los científicos a rastrear la historia del virus del sarampión y a situar su origen en el siglo VI antes de Cristo.
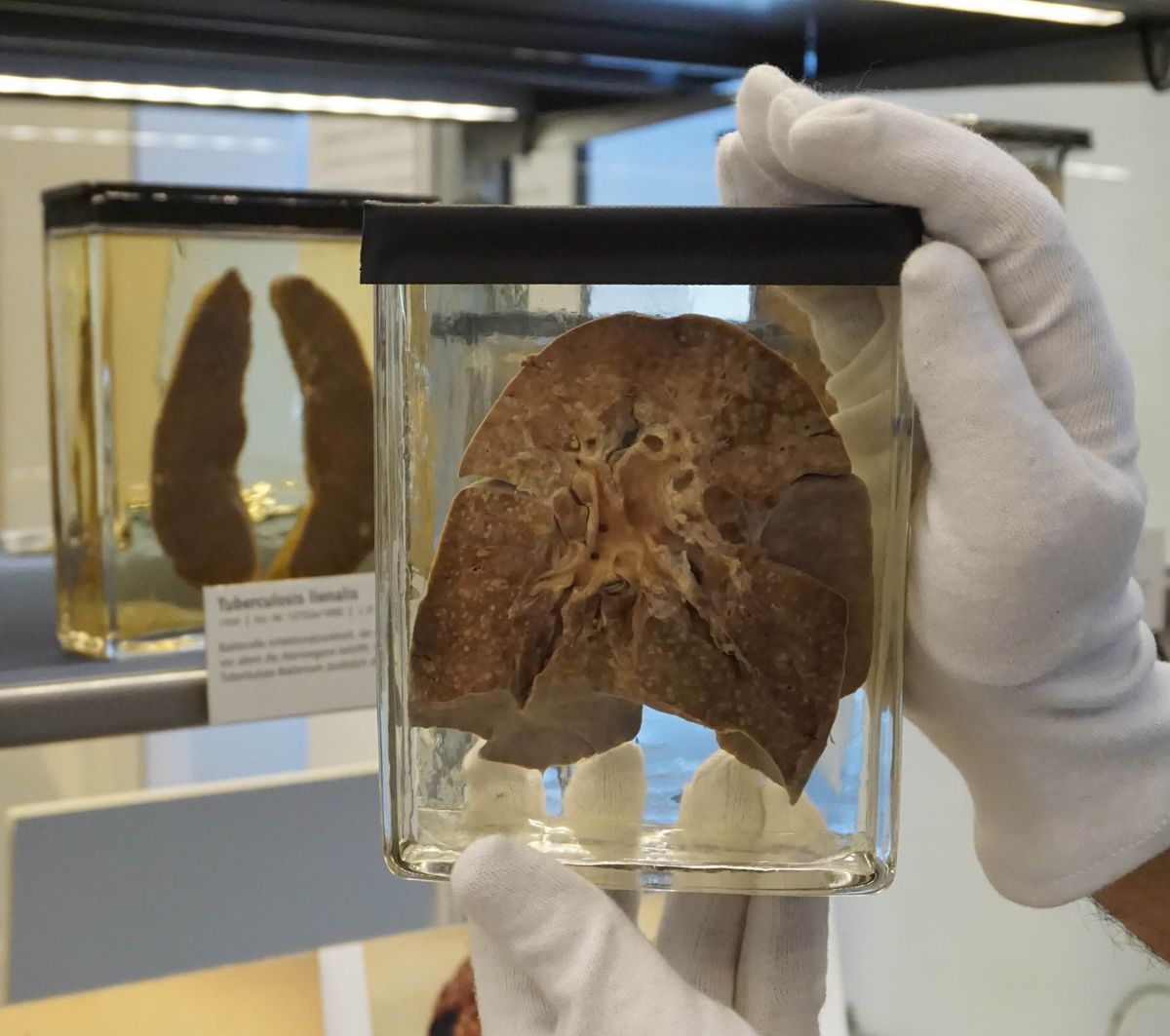
Durante años, el pulmón permaneció en el sótano del Museo de Historia de la Medicina de Berlín junto con otros cientos de especímenes pulmonares, todos ellos recogidos y conservados entre los años 1870 y 1930. A la caza de patógenos respiratorios bien conservados, el virólogo Sébastien Calvignac-Spencer, del Instituto Robert Koch, y su equipo de investigación bajaron al sótano y se asomaron a todos y cada uno de los frascos. «Es una cuestión de serendipia» que el equipo encontrara un pulmón perteneciente a un paciente de sarampión de 2 años que murió de la enfermedad en 1912, dijo Calvignac-Spencer.
El equipo logró extraer muestras del virus del tejido pulmonar de 108 años de antigüedad y utilizó el material genético -el genoma del sarampión más antiguo jamás secuenciado- para aprender más sobre los orígenes del patógeno. En un nuevo estudio, publicado hoy (18 de junio) en la revista Science, estiman que el sarampión podría haber divergido de su pariente más cercano conocido, un virus bovino ya erradicado, en una fecha tan temprana como el año 528 a.C.
La nueva estimación sugiere que el virus puede ser «más de mil años más antiguo que cualquier otra estimación anterior», dijo Calvignac-Spencer a Live Science.
Relación: 11 enfermedades (a veces) mortales que saltaron entre especies
Un raro hallazgo
Estudios anteriores predecían que el sarampión y el extinto virus del ganado, llamado peste bovina, se separaron de su ancestro común más reciente entre los siglos XI y XII, según un informe de 2011 publicado en la revista Molecular Biology and Evolution (MBE). Sin embargo, el médico persa Muhammad ibn Zakariya al-Razi escribió una descripción clínica del sarampión en el siglo X, así que algo no cuadraba.
«La división entre el sarampión y la peste bovina está claramente subestimada», dijo Joel Wertheim, autor del informe de la MBE y profesor adjunto de medicina en la Universidad de California, San Diego, que no participó en el nuevo estudio de Science. Estas subestimaciones se deben a dos problemas críticos: la falta de muestras antiguas de sarampión y las suposiciones erróneas sobre cómo muta el virus a lo largo del tiempo, que desvían los modelos evolutivos hacia una «fecha ridículamente reciente», dijo Wertheim a Live Science.
Wertheim y sus coautores construyeron un nuevo modelo para tener en cuenta estos factores y retrasaron la fecha de origen hasta finales del siglo IX, pero «no creímos estar en lo cierto», dijo. Ahora, Calvignac-Spencer y su equipo han llegado a una estimación más realista, en parte, al incluir en su análisis el espécimen recién descubierto en 1912, dijo Wertheim.
Relacionado: Volviéndose viral: 6 nuevos descubrimientos sobre los virus

Antes de que el equipo encontrara la muestra de 1912, el genoma del sarampión más antiguo secuenciado databa de 1954, señalaron los autores. Los científicos calculan la tasa de cambio evolutivo, es decir, cuánto y con qué rapidez muta un virus, comparando muestras recogidas en distintos momentos y rastreando las diferencias en su código genético. Cuantas más muestras y más antiguas examinemos, más claro será ese ritmo de cambio, dijo Calvignac-Spencer.
Pero la columna vertebral del virus del sarampión es el ARN, un tipo de material genético que se degrada rápidamente en comparación con su primo más resistente, el ADN. La muestra de 1912 escapó a este destino porque el pulmón había sido fijado en formalina, un conservante que detiene las reacciones químicas que de otro modo degradarían el ARN. La formalina también «pega» el ARN conservado a las moléculas cercanas, lo que dificulta su extracción, explicó Calvignac-Spencer.
Para despegar el ARN, el equipo cortó 200 miligramos de tejido del pulmón e hirvió la diminuta muestra, lo que hizo que las moléculas pegajosas de su interior se desprendieran sin destruir el ARN. A continuación, el equipo construyó un genoma «casi completo» a partir del ARN rescatado, escribieron. Para enriquecer aún más su modelo evolutivo, el equipo recorrió la colección de muestras genéticas del Laboratorio Nacional de Referencia de Alemania y encontró dos muestras de sarampión recogidas en 1960 para añadirlas a su análisis.
Construyendo mejores modelos
El equipo construyó su modelo evolutivo a partir de la muestra de 1912, las muestras de 1960 y 127 muestras adicionales, la mayoría recogidas en la década de 1990 o después. Un segundo modelo comparó unas 50 secuencias de sarampión con el virus de la peste bovina, que se declaró erradicado en 2011, y con su pariente más cercano, el virus de la peste de pequeños rumiantes (PPRV), que infecta a cabras y ovejas, para precisar cuándo se separaron estos patógenos de su ancestro común.
En ambos modelos, el equipo tuvo en cuenta un fenómeno llamado «selección purificadora», que muchos estudios anteriores pasaron por alto, dijo Calvignac-Spencer. Mientras que algunas presiones evolutivas añaden mutaciones útiles al genoma y lo mantienen estable a lo largo del tiempo, la llamada selección purificadora purga las mutaciones perjudiciales del genoma antes de que puedan acumularse. Estas fuerzas complementarias ayudan a establecer el ritmo del cambio evolutivo, por lo que para estimar cuándo surgió el sarampión por primera vez, hay que tener en cuenta la selección purificadora, dijo Wertheim.
«Se puede cambiar en un orden de magnitud si se tiene en cuenta la selección purificadora», dijo. La selección purificadora, en parte, hace que ciertos segmentos del genoma muten con facilidad y frecuencia, mientras que otros apenas cambian, añadió. «Habrá varias mutaciones que golpeen la misma posición una y otra vez», pero como sólo se tiene un número limitado de muestras, es posible que se pasen por alto algunas de esas mutaciones, dijo Calvignac-Spencer. El equipo diseñó su modelo para captar estas mutaciones que, de otro modo, podrían pasar desapercibidas.
Según el momento en que divergieron la peste bovina y el sarampión, la «fecha más temprana posible para el establecimiento del sarampión en las poblaciones humanas» se produjo alrededor del siglo VI a.C., aunque la fecha exacta en que el virus infectó por primera vez a las personas sigue siendo desconocida.
Los autores señalaron que, hace entre 2.000 y 2.500 años, los seres humanos empezaron a construir asentamientos lo suficientemente grandes como para sostener un brote de sarampión, ofreciendo al virus la oportunidad de establecerse. El sarampión tiende a desaparecer en comunidades de menos de 250.000 personas, ya que los residentes se vuelven rápidamente inmunes a la enfermedad o mueren a causa de ella, por lo que «las poblaciones humanas pequeñas sólo podrían servir como huéspedes sin salida», escribieron.
Calvignac-Spencer dijo que está interesado en descubrir muestras antiguas de sarampión, si es que existen, con el fin de perfeccionar nuestra comprensión de la historia del patógeno. Wertheim dijo que predice que más virólogos se unirán a la caza de muestras antiguas que acechan en los sótanos de los museos y en los archivos de los hospitales.
«Me quedé asombrado cuando vi que eran capaces de extraer un virus de más de 100 años del tejido pulmonar», dijo Wertheim. Creo que más virólogos empezarán a utilizar «virus cada vez más antiguos a medida que la gente sea más ambiciosa y se anime con estos resultados», añadió.
- 20 de las peores epidemias y pandemias de la historia
- Los 12 virus más mortíferos de la Tierra
- Las 10 enfermedades más misteriosas
Originalmente publicado en Live Science.

¡Oferta: ahorre un 45% en ‘Cómo funciona’ ‘Todo sobre el espacio’ y ‘Todo sobre la historia’!
Por tiempo limitado, puede contratar una suscripción digital a cualquiera de nuestras revistas científicas más vendidas por sólo 2 dólares.38 al mes, o un 45% de descuento sobre el precio estándar durante los tres primeros meses.Ver Oferta
Noticias recientes
