- Genoma Sequenciação
- Caracterização genética
- Métodos de sequenciação do genoma da gripe
Sequenciação do genoma
Vírus da gripe estão em constante mudança, Na verdade, todos os vírus da gripe sofrem alterações genéticas ao longo do tempo (para mais informações, veja Como o Vírus da Gripe Pode Mudar: “Deriva” e “Shift”). O genoma de um vírus da gripe consiste em todos os genes que compõem o vírus. O CDC realiza vigilância durante todo o ano dos vírus da gripe circulante para monitorar as mudanças no genoma (ou partes do genoma) desses vírus. Este trabalho é realizado como parte da rotina de vigilância da gripe dos EUA e como parte do papel do CDC como Centro Colaborador da Organização Mundial de Saúde (OMS) para Referência e Pesquisa sobre a Influenza. A informação que o CDC coleta do estudo das mudanças genéticas (também conhecidas como “substituições”, “variantes” ou “mutações”) dos vírus da influenza desempenha um papel importante na saúde pública, ajudando a determinar se vacinas e medicamentos antivirais funcionarão contra os vírus da influenza que circulam atualmente, além de ajudar a determinar o potencial dos vírus da influenza em animais para infectar seres humanos.
O sequenciamento genético revela a seqüência dos nucleotídeos em um gene, como letras do alfabeto em palavras. Os nucleotídeos são moléculas orgânicas que formam o bloco estrutural de construção de unidade de ácidos nucléicos, como RNA ou DNA. Todos os vírus de influenza consistem de RNA de cadeia única em oposição a DNA de cadeia dupla. Os genes de RNA de vírus de influenza são feitos de cadeias de nucleotídeos que são unidos e codificados pelas letras A, C, G e U, que significam adenina, citosina, guanina, e uracil, respectivamente. Comparar a composição dos nucleotídeos em um gene de vírus com a ordem dos nucleotídeos em um gene de vírus diferente pode revelar variações entre os dois vírus.
Variações genéticas são importantes porque podem afetar a estrutura das proteínas de superfície de um vírus da influenza. As proteínas são feitas de sequências de aminoácidos.
A substituição de um aminoácido por outro pode afectar as propriedades de um vírus, tais como a forma como um vírus se transmite entre as pessoas, e como o vírus é susceptível a medicamentos antivirais ou vacinas actuais.
 >464646>
>464646>
 >464646>
>464646>
O sequenciamento genético revela a seqüência dos nucleotídeos em um gene, como letras do alfabeto em palavras. Comparar a composição dos nucleotídeos em um gene de vírus com a ordem dos nucleotídeos em um gene de vírus diferente pode revelar variações entre os dois vírus.
Variações genéticas são importantes porque afetam a estrutura das proteínas de superfície de um vírus da influenza. As proteínas são feitas de sequências de aminoácidos.
A substituição de um aminoácido por outro pode afectar as propriedades de um vírus, tais como a forma como um vírus se transmite entre as pessoas, e como o vírus é susceptível a medicamentos antivirais ou vacinas actuais.
Vírus influenza A e B – os vírus influenza primários que infectam as pessoas – são vírus RNA que têm oito segmentos genéticos. Estes genes contêm ‘instruções’ para fazer novos vírus, e são estas instruções que um vírus influenza usa quando infecta uma célula humana para enganar a célula para produzir mais vírus influenza, espalhando assim a infecção.
>
Gens do Influenza consistem de uma seqüência de moléculas chamadas nucleotídeos que se ligam em forma de cadeia. Os nucleotídeos são designados pelas letras A, C, G e U.
O sequenciamento genético é um processo que determina a ordem, ou sequência, dos nucleotídeos (isto é, A, C, G e U) em cada um dos genes presentes no genoma do vírus. O sequenciamento completo do genoma pode revelar a sequência de aproximadamente 13.500 letras de todos os genes do genoma do vírus.
Cada ano o CDC executa o sequenciamento completo do genoma em cerca de 7.000 vírus da gripe a partir de amostras clínicas originais recolhidas através da vigilância virológica. Um genoma do vírus influenza A ou B contém oito segmentos de genes que codificam (ou seja, determinam a estrutura e características das 12 proteínas do vírus, incluindo suas duas proteínas primárias de superfície: hemaglutinina (HA) e neuraminidase (NA). As proteínas de superfície de um vírus influenza determinam propriedades importantes do vírus, incluindo como o vírus responde a certos medicamentos antivirais, a similaridade genética do vírus com os vírus da vacina atual da influenza e o potencial dos vírus zoonóticos (de origem animal) da influenza para infectar hospedeiros humanos.
Caracterização Genética
CDC e outros laboratórios de saúde pública ao redor do mundo têm sequenciado os genes dos vírus influenza desde os anos 80. O CDC contribui com seqüências de genes para bases de dados públicas, como o ícone GenBankexternal e o ícone externo da Iniciativa Global de Compartilhamento de Dados sobre Influenza Aviária (GISAID), para uso por pesquisadores de saúde pública. As bibliotecas de seqüências de genes resultantes permitem ao CDC e a outros laboratórios comparar os genes dos vírus da influenza atualmente em circulação com os genes dos vírus da influenza mais antigos e os vírus usados em vacinas. Este processo de comparação de seqüências genéticas é chamado de caracterização genética. O CDC usa a caracterização genética pelas seguintes razões:
- Para determinar o quão “relacionados” ou similares os vírus da gripe estão geneticamente
- Para monitorar como os vírus da gripe estão evoluindo
- Para identificar alterações genéticas que afetam as propriedades do vírus. Por exemplo, para identificar as mudanças específicas que estão associadas aos vírus da gripe que se propagam mais facilmente, causando doenças mais graves, ou desenvolvendo resistência a medicamentos antivirais
- Para avaliar o quão bem uma vacina contra a gripe poderia proteger contra um determinado vírus da gripe com base na sua semelhança genética com o vírus
- Para monitorar mudanças genéticas nos vírus da gripe que circulam em populações animais que poderiam infectar humanos.
As diferenças relativas entre um grupo de vírus da gripe são mostradas organizando-os em um gráfico chamado ‘árvore filogenética’. As árvores filogenéticas para os vírus da gripe são como árvores familiares (genealogia) para as pessoas. Estas árvores mostram o quanto os vírus individuais estão ‘relacionados’ uns com os outros. Os vírus são agrupados com base no facto de os nucleótidos dos seus genes serem idênticos ou não. As árvores filogenéticas dos vírus da influenza geralmente mostram quão semelhantes os genes da hemaglutinina (HA) ou neuraminidase (NA) dos vírus são uns aos outros. Cada sequência de um vírus específico da gripe tem o seu próprio ramo na árvore. O grau de diferença genética (número de diferenças nucleotídicas) entre os vírus é representado pelo comprimento das linhas horizontais (ramos) na árvore filogenética. Quanto mais afastados estiverem os vírus no eixo horizontal de uma árvore filogenética, mais geneticamente diferentes os vírus são entre si.
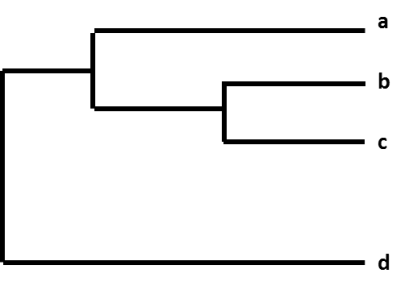
Figure. Uma árvore filogenética.
Por exemplo, após sequências de CDC um vírus influenza A(H3N2) coletado através de vigilância, a sequência de vírus é catalogada com outras sequências de vírus que têm um gene HA (H3) similar, e um gene NA similar (N2). Como parte deste processo, a CDC compara a nova sequência de vírus com as outras sequências de vírus, e procura diferenças entre elas. O CDC então usa uma árvore filogenética para representar visualmente quão geneticamente diferentes os vírus A(H3N2) são uns dos outros.
CDC realiza a caracterização genética dos vírus da influenza o ano todo. Estes dados genéticos são usados em conjunto com dados de caracterização antigênica de vírus para ajudar a determinar quais vírus de vacinas devem ser escolhidos para as próximas vacinas contra influenza do hemisfério norte ou do hemisfério sul. Nos meses que antecedem as reuniões de consulta da OMS em fevereiro e setembro, o CDC coleta os vírus da influenza através da vigilância e compara as seqüências gênicas HA e NA dos vírus vacinais atuais com as dos vírus da gripe em circulação. Esta é uma forma de avaliar quão estreitamente relacionados os vírus da gripe circulante estão com os vírus contra os quais a vacina da gripe sazonal foi formulada para se proteger. Como os vírus são coletados e caracterizados geneticamente, as diferenças podem ser reveladas.
Por exemplo, às vezes, ao longo de uma estação, os vírus circulantes mudam geneticamente, o que faz com que eles se tornem diferentes do vírus vacinado correspondente. Esta é uma indicação de que um vírus vacinal diferente pode precisar ser selecionado para a vacina da próxima estação de gripe, embora outros fatores, incluindo os achados da caracterização antigênica, influenciem fortemente as decisões sobre a vacina. As proteínas de superfície HA e NA dos vírus da gripe são antígenos, o que significa que são reconhecidos pelo sistema imunológico e são capazes de desencadear uma resposta imunológica, incluindo a produção de anticorpos que podem bloquear a infecção. A caracterização antigénica refere-se à análise da reacção de um vírus com anticorpos para ajudar a avaliar a sua relação com outro vírus.
Métodos de Sequenciação do Genoma da Gripe
Uma amostra de gripe contém muitas partículas de vírus da gripe que foram cultivadas num tubo de ensaio e que muitas vezes têm pequenas diferenças genéticas em comparação umas com as outras entre toda a população de vírus irmãos.
Tradicionalmente, os cientistas têm usado uma técnica de sequenciamento chamada “a reação Sanger” para monitorar a evolução da gripe como parte da vigilância virológica. O sequenciamento de Sanger identifica a seqüência genética predominante entre os muitos vírus da influenza encontrados em um isolado. Isto significa que pequenas variações na população de vírus presentes em uma amostra não são refletidas no resultado final. Os cientistas frequentemente usam o método Sanger para conduzir o sequenciamento genético parcial dos vírus da influenza, enquanto as novas tecnologias (ver próximo parágrafo) são mais adequadas para o sequenciamento genético completo.
Nos últimos cinco anos, o CDC tem usado metodologias de “Next Generation Sequencing (NGS)”, que expandiram muito a quantidade de informações e detalhes que a análise do sequenciamento pode fornecer. O NGS usa a detecção molecular avançada (AMD) para identificar seqüências de genes de cada vírus em uma amostra. Portanto, o NGS revela as variações genéticas entre muitas partículas diferentes do vírus influenza em uma única amostra, e estes métodos também revelam toda a região codificadora dos genomas. Este nível de detalhe pode beneficiar diretamente a tomada de decisões de saúde pública de formas importantes, mas os dados devem ser cuidadosamente interpretados por especialistas altamente treinados, no contexto de outras informações disponíveis. Ver Projetos AMD: Improving Influenza Vaccines para mais informações sobre como NGS e AMD estão revolucionando o mapeamento do genoma da gripe no CDC.
