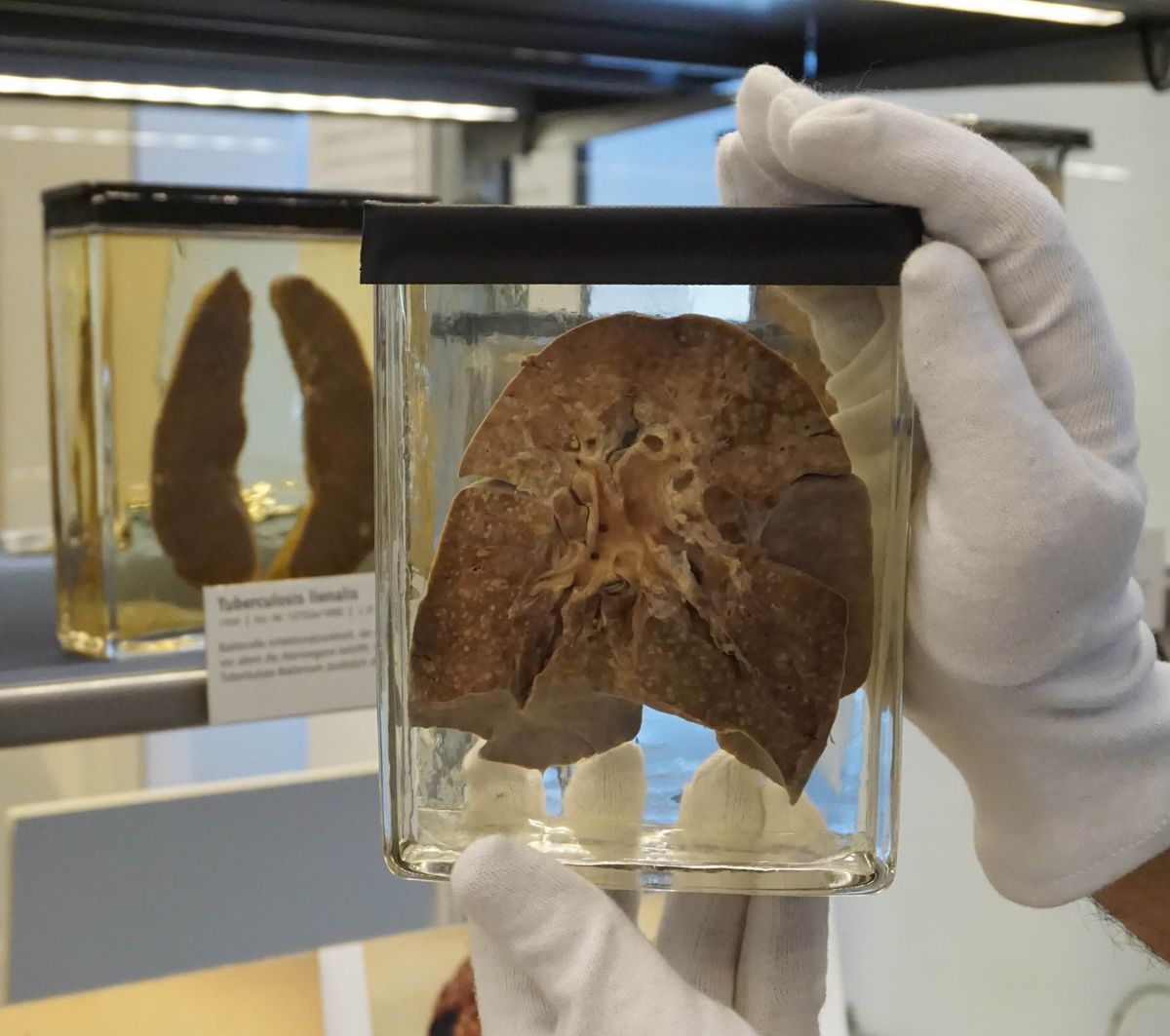
Um pulmão humano doente, fixado no conservante formalina por mais de 100 anos, ajudou os cientistas a rastrear a história do vírus do sarampo e a colocar sua origem até o século VI a.C.
Durante anos, o pulmão sentou-se no porão do Museu de História Médica de Berlim junto com centenas de outros espécimes de pulmão, todos coletados e preservados entre os anos 1870 e 1930. Numa caça aos patógenos respiratórios bem preservados, o virologista Sébastien Calvignac-Spencer do Instituto Robert Koch e a sua equipa de investigação desceram ao porão e espreitaram em cada um dos frascos. “É uma questão de serendipidade” que a equipe encontrou um pulmão pertencente a um paciente de 2 anos com sarampo que morreu da doença em 1912, disse Calvignac-Spencer.
A equipe conseguiu extrair amostras do vírus do tecido pulmonar de 108 anos e usou o material genético – o genoma mais antigo já sequenciado – para aprender mais sobre as origens do patógeno. Em um novo estudo, publicado hoje (18 de junho) na revista Science, eles estimam que o sarampo poderia ter divergido de seu parente mais próximo conhecido, um vírus bovino agora erradicado, já em 528 a.C.
A nova estimativa sugere que o vírus pode ser “mais de 1.000 anos mais velho do que qualquer estimativa anterior”, disse Calvignac-Spencer à Live Science.
Relacionado: 11 (às vezes) doenças mortais que saltaram entre as espécies
Uma descoberta rara
Estudos anteriores previram que o sarampo e o vírus do gado extinto, chamado peste bovina, se separaram de seus ancestrais comuns mais recentes entre os séculos 11 e 12, de acordo com um relatório de 2011 na revista Molecular Biology and Evolution (MBE). No entanto, o médico persa Muhammad ibn Zakariya al-Razi escreveu uma descrição clínica do sarampo no século 10, por isso algo não bateu certo.
“A divisão entre sarampo e peste bovina é claramente subestimada”, disse Joel Wertheim, autor do relatório MBE e professor assistente de medicina na Universidade da Califórnia, San Diego, que não estava envolvido no novo estudo científico. Essas subestimações surgem de duas questões críticas: a falta de amostras antigas de sarampo e suposições errôneas sobre como o vírus mutua através do tempo, que enviesam modelos evolutivos em direção a uma “data ridiculamente recente”, disse Wertheim ao Live Science.
Wertheim e seus coautores construíram um novo modelo para explicar esses fatores e empurraram a data de origem para o final do século IX, mas “não achamos que estávamos certos”, disse ele. Agora, Calvignac-Spencer e sua equipe chegaram a uma estimativa mais realista, em parte, ao incluir o recém-descoberto exemplar de 1912 em suas análises, disse Wertheim.
Relacionado: Going viral: 6 novas descobertas sobre vírus

Antes de a equipe encontrar a amostra de 1912, o genoma mais antigo já sequenciado, datado de 1954, os autores observaram. Os cientistas estimam a taxa de mudança evolutiva, ou quanto e quão rapidamente um vírus se altera, comparando amostras coletadas em diferentes momentos e rastreando diferenças em seu código genético. Quanto mais e mais velhas amostras examinamos, mais clara se torna essa taxa de mudança, disse Calvignac-Spencer.
Mas a espinha dorsal do vírus do sarampo é o RNA, um tipo de material genético que se degrada rapidamente em comparação com o DNA de seu primo mais resistente. A amostra de 1912 escapou deste destino porque o pulmão tinha sido fixado em formalina, um conservante que interrompe reações químicas que de outra forma degradariam o RNA. Formalina também “cola” o RNA preservado às moléculas próximas, tornando difícil de extrair, Calvignac-Spencer disse.
Para descolar o RNA, a equipe cortou 0.007 onças (200 miligramas) de tecido do pulmão e ferveu a amostra minúscula, fazendo com que as moléculas pegajosas dentro se desfizessem sem destruir o RNA. A equipe então construiu “um genoma quase completo” a partir do RNA resgatado, eles escreveram. Para enriquecer ainda mais seu modelo evolutivo, a equipe vasculhou a coleta de amostras genéticas no Laboratório Nacional de Referência Alemão e encontrou duas amostras de sarampo coletadas em 1960 para adicionar à sua análise.
Construindo melhores modelos
A equipe construiu seu modelo evolutivo a partir da amostra de 1912, amostras de 1960 e 127 amostras adicionais, a maioria coletada em ou após os anos 90. Um segundo modelo comparou cerca de 50 sequências de sarampo com o vírus da peste bovina, que foi declarado erradicado em 2011, e a sua peste relativa mais próxima dos pequenos ruminantes (PPRV), que infecta caprinos e ovinos, para identificar quando estes patogénios se separaram dos seus antepassados comuns.
Em ambos os modelos, a equipe levou em conta um fenômeno chamado “seleção purificadora”, que muitos estudos anteriores ignoraram, disse Calvignac-Spencer. Enquanto algumas pressões evolucionárias adicionam mutações úteis ao genoma, e o mantêm estável ao longo do tempo, a chamada seleção purificadora purga mutações prejudiciais do genoma antes que elas possam se acumular. Essas forças complementares ajudam a definir o ritmo da mudança evolucionária, portanto, para estimar quando o sarampo surgiu pela primeira vez, você deve levar em conta a seleção purificadora, disse Wertheim.
“Você pode mudar por uma ordem de magnitude levando em conta a seleção purificadora”, disse ele. A seleção purificadora, em parte, faz com que certos segmentos do genoma sofram mutações rápidas e frequentes, enquanto outros quase não mudam nada, acrescentou ele. “Você terá várias mutações atingindo a mesma posição repetidamente”, mas como você tem apenas um número limitado de amostras, você pode perder algumas dessas mutações, disse Calvignac-Spencer. A equipe projetou seu modelo para capturar essas mutações que podem faltar.
Baseado em quando a peste bovina e o sarampo divergiram, a “data mais próxima possível para o estabelecimento do sarampo nas populações humanas” ocorreu por volta do século sexto a.C., embora a data exacta em que o vírus infectou as primeiras pessoas permaneça desconhecida.
Os autores observaram que, há cerca de 2.000 a 2.500 anos, os humanos começaram a construir povoações suficientemente grandes para sustentar um surto de sarampo, oferecendo ao vírus uma oportunidade de se estabelecer. O sarampo tende a desaparecer em comunidades de menos de 250.000 indivíduos, pois os residentes rapidamente se tornam imunes à doença ou morrem dela, de modo que “pequenas populações humanas só poderiam servir como hospedeiros sem saída”, escreveram eles.
Calvignac-Spencer disse que está interessado em descobrir amostras de sarampo antigo, se elas existirem, a fim de refinar ainda mais a nossa compreensão da história do patógeno. Wertheim disse que ele prevê que mais virologistas se juntarão à caça de amostras antigas à espreita em porões de museus e arquivos hospitalares.
“Fiquei surpreso quando vi que eles foram capazes de retirar um vírus com mais de 100 anos do tecido pulmonar”, disse Wertheim. Acho que mais virologistas começarão a usar “vírus cada vez mais antigos à medida que as pessoas se tornam mais ambiciosas e encorajadas por esses resultados”, acrescentou ele.
- 20 das piores epidemias e pandemias da história
- Os 12 vírus mais mortais da Terra
- As 10 doenças mais misteriosas
Originalmente publicado em Live Science.

OFFER: Economize 45% em ‘Como Funciona’ ‘Tudo Sobre o Espaço’ e ‘Tudo Sobre a História’!
Por um tempo limitado, você pode fazer uma assinatura digital de qualquer uma de nossas revistas de ciência mais vendidas por apenas $2.Ver negócio
Recent news
